Hemos estudiado los genes supresores de tumores PTEN y p16 a modo comparativo
en 22 astrocitomas de adultos de alto grado (glioblastomas, GBMs) y 34 astrocitomas
pediatricos de bajo grado. La extraccion de DNA de los GBMs se realizó a partir
de muestras congeladas, mientras que los astrocitomas pediátricos correspondían
a piezas de archivo, incluidas en parafina.
Los 9 exones del gen PTEN fueron amplificados mediante PCR y sometidos a SSCP
para detectar posibles mutaciones en su secuencia. Se utilizaron geles verticales
de 10X8 cm, de poliacrilamida al 12%, con 5% de glicerol, en condiciones no
desnaturalizantes, a temperatura ambiente, con recirculacion de agua. Posteriormente,
los casos positivos se sometieron a secuenciacion. Se detectaron 5 mutaciones
en un total de 22 GBMs (23%) (Fig. 1).
Tambien se analizaron las deleciones homocigoticas de PTEN mediante PCR diferencial,
utilizando oligonucleotidos para PTEN junto a oligonucleotidos para el gen GADPH.
Se detectaron 3 deleciones homocigoticas entre los 22 GBMs (14%) (Fig.
2).
Hasta el momento hemos estudiado las posibles mutaciones (no las deleciones
homocigoticas) de PTEN en 34 astrocitomas pediatricos de bajo grado. Tras haber
analizado 5 de los 9 exones del gen, no se ha demostrado ninguna mutacion.
El gen p16 se estudio en 21 GBMs, no revelando mutacion alguna en sus 3 exones.
Sin embargo, 6 GBMs de 21 (29%) presentaron deleciones homocigoticas de p16
(Fig. 3). Tambien se demostro, mediante
MSP (methylation specific PCR) que el promotor de p16 sufria hipermetilacion
en 2 de los 21 GBMs (9%) (Fig. 4).
Hasta el momento hemos estudiado el gen p16 en 34 astrocitomas pediatricos
de bajo grado: no se han detectado mutaciones en la mayor parte de la secuencia
exonica de p16. Respecto al posible papel de las deleciones homocigoticas en
este tipo de astrocitomas, la baja calidad del DNA extraido de parafina ha hecho
muy dificil la objetivacion de las mismas, por lo que en este momento se trabaja
en el diseño de un buen protocolo de estudio de deleciones homocigoticas de
p16 a partir de muestras incluidas en parafina.
Tras estos estudios concluimos que PTEN muestra alteraciones (mutaciones o
deleciones homocigoticas) en un 32% de GBMs (7 de 22 casos), mientras que p16
sufre deleciones homocigoticas o hipermetilacion de su promotor en un 38% de
GBMs (8 de 21 casos) (Tabla 1). El estudio
de las posibles alteraciones de PTEN mediante deleciones homocigoticas, asi
como las de p16 (deleciones homocigoticas e hipermetilacion del promotor) no
ha sido finalizado aun en los tumores pediatricos.
FIGURAS
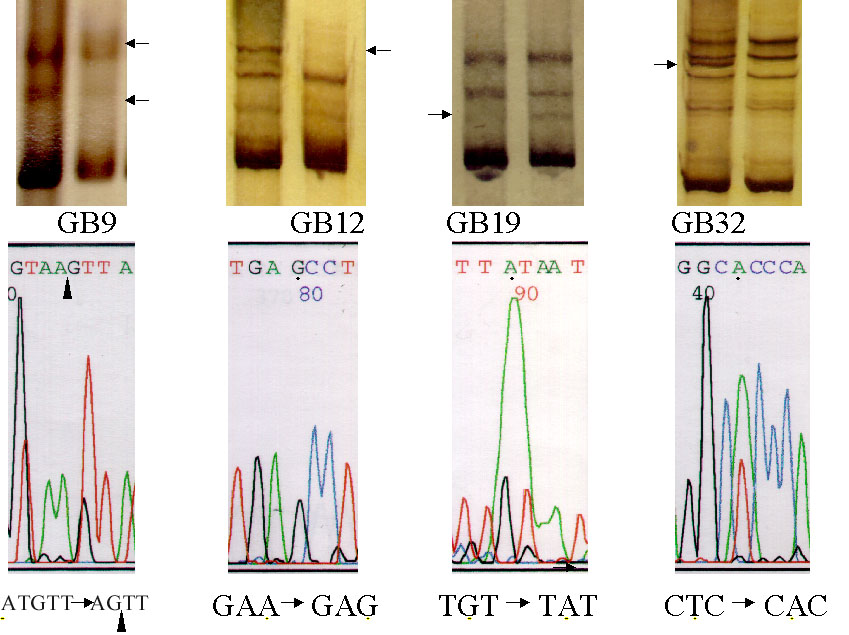
Figura 1. Resultados de SSCP y secuenciación de diferentes
exones de PTEN en 4 casos de glioblastoma multiforme.
|
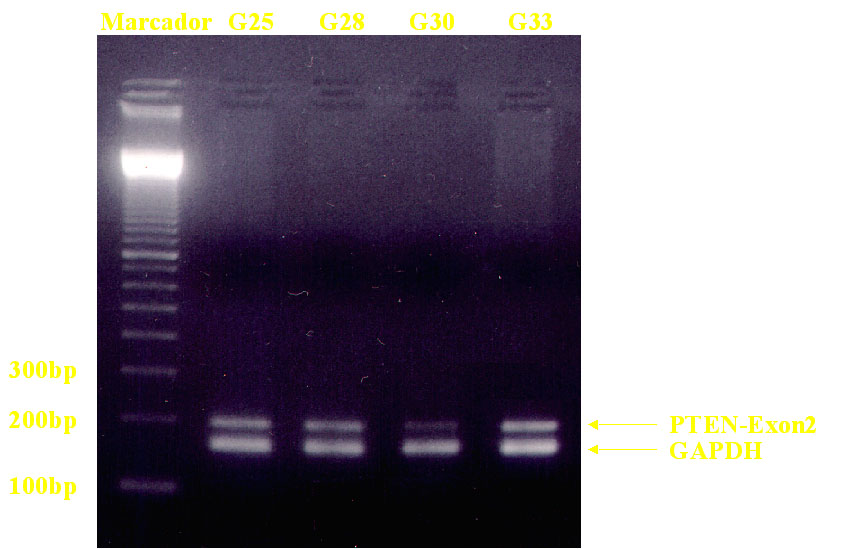
Figura 2. Deleción homocigótica de PTEN demostrada en
un glioblastoma (G30). Gel de agarosa al 2% teñido con bromuro de
etidio tras ensayo de PCR diferencial para detectar deleción homocigótica
de PTEN. Cada uno de los DNA extraídos de cuatro glioblastomas muestra
dos bandas: la superior corresponde a un fragmento del exón 2 del gen
PTEN de 202 bp, y la banda inferior corresponde a un fragmento de 160
pb del gen GAPDH. Se consideró deleción homocigótica del exón 2 de PTEN
cuando la banda de GAPDH que representa el DNA normal era 3 veces más
intensa. Para la determinación del tamaño se incluyó en la primera calle
un marcador con bandas de tamaño creciente en 100 pb (Pharmacia Biotech).
|
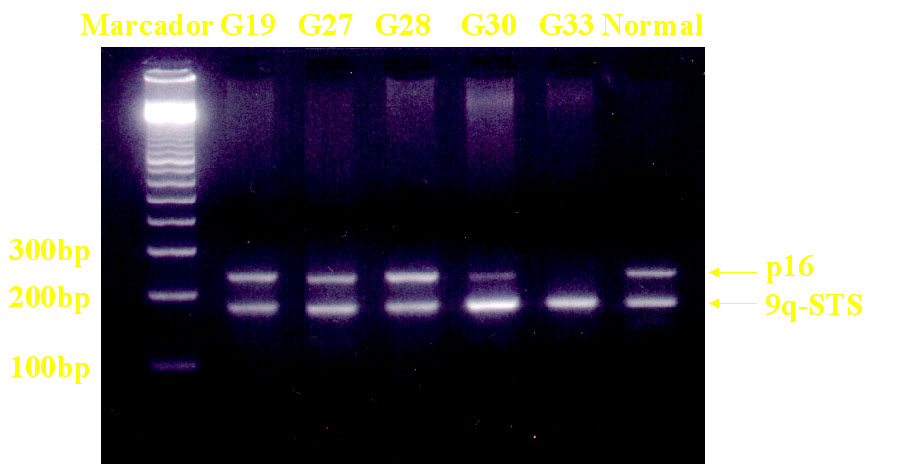
Figura 3. Deleción homocigótica de p16 demostrada en dos glioblastomas
(G30 y G33). Gel de agarosa al 2% teñido con bromuro de etidio
tras ensayo de PCR diferencial para detectar deleción homocigótica de
p16. Cada uno de los DNA extraídos de cinco glioblastomas y leucocitos
de donantes sanos, muestra dos bandas: la superior corresponde a un
fragmento del exón 2 del gen p16 de 235 pb, y la banda inferior corresponde
a un fragmento de 180 pb de 9qSTS. Se consideró deleción homocigótica
del exón 2 de p16 cuando la banda de STS que representa el DNA normal
era 3 veces más intensa. Para la determinación del tamaño se incluyó
en la primera calle un marcador con bandas de tamaño creciente en 100
pb (Pharmacia Biotech).
|
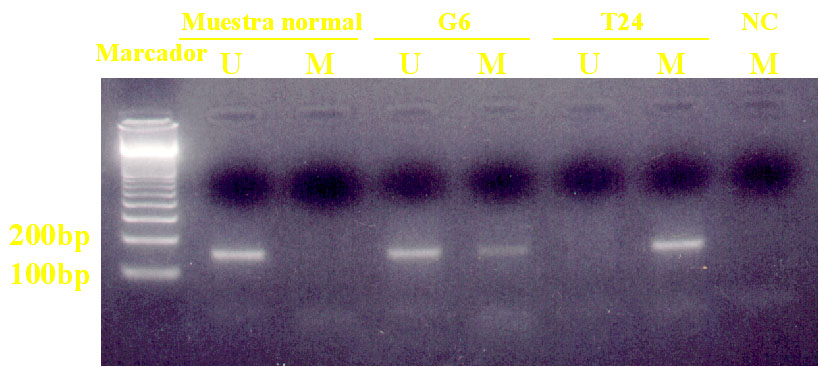
Figura 4. Inactivación de p16 por hipermetilación del
promotor en un glioblastoma (G6). Gel de agarosa al 2% teñido con
bromuro de etidio tras ensayo de PCR diferencial para detectar regiones
del promotor de p16 no metiladas (U) o metiladas (M). Muestra normal:
DNA de leucocitos, usado como control positivo de promotor de p16 no
metilado. El DNA de la línea celular T24 se usó como control positivo
de metilación en p16. NC: PCR sin DNA, usado como control negativo de
la reacción de PCR. Para la determinación del tamaño se incluyó en la
primera calle un marcador con bandas de tamaño creciente en 100 pb (Pharmacia
Biotech).
|
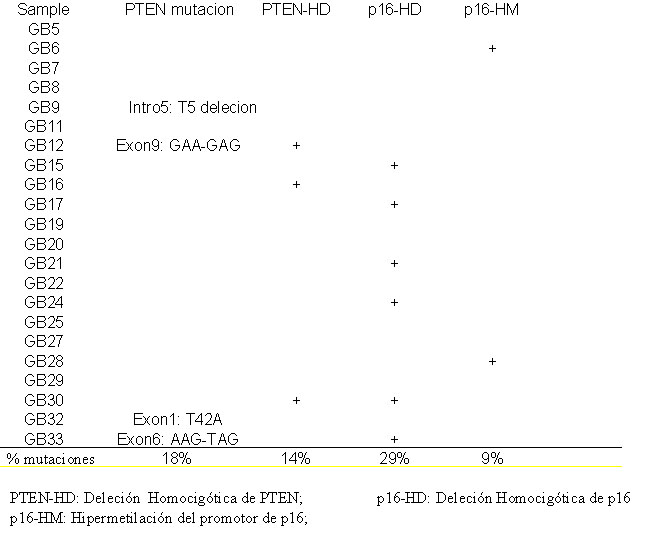
Tabla1: Alteraciones genéticas de PTEN, p16 y CDK4 en glioblastoma
multiforme
|




